
こんにちは!データサイエンティストの青木和也(https://twitter.com/kaizen_oni)です!
この記事では、「APROACHING (ALMOST) ANY MACHINE LEARNING PROBLEM」のコード部分で、Pythonや他ライブラリの仕様変更に伴ってコードが上手くいかない部分について、2024/1/17現在のバージョンに基づいたコードの修正版を掲載していきたいと思います。
私自身もコードが上手くいかずになんでだろう?と苦戦したところなので、他にも同書籍を学習中の方の一助になれば幸いです!
P11のMNISTのイメージ画像を出力するコード
修正版のコードは以下になります。
single_image = pixel_values.iloc[1, :].values.reshape(28,28)
plt.imshow(single_image, cmap = 'gray')pixel_valuesはpandasのDataFrameですが、元コードだとDataFrameには不適切な範囲選択の仕方をしているので、ここはilocを使用してDataFrameに適した範囲指定の仕方をとります。
また、DataFrameに対してreshapeは適用できないので、valuesを間に入れることによって配列形式に直してあげ、その後reshapeを使用して28*28の行列に変換します。
参考ブログ: データフレームのデータスライス時に発生するエラー
P12のMNISTをt-SNEで変換するコード
修正版のコードは以下になります。
tsne = manifold.TSNE(n_components=2, random_state = 42)
transformed_data = tsne.fit_transform(pixel_values.iloc[:3000, :])ここも上の修正と同様にilocを使用して、DataFrameにあった範囲指定の方法に変更してあげます。
P13のt-SNEで変換したMNISTデータを2次元プロットするコード
修正版のコードは以下になります。
grid = sns.FacetGrid(tsne_df, hue = 'targets', height = 8)
grid.map(plt.scatter, 'x', 'y').add_legend()2024年時点でのSeabornのFacetGridの__init__メソッドにはsizeという引数は存在しないので、sizeの代わりにheightを指定して、プロットの高さを指定してあげます。
P122~123のロジスティック回帰分析
修正版のコードは以下になります。
def run(fold):
#load ...
df = pd.read_csv('../input/adult_folds.csv')
#####################
中略
#####################
for col in features:
df.loc[:, col] = df[col].astype(str).fillna('NONE')
# 以下のコードを挿入
df.income = df.income.astype(int)
# get...
df_train = df[df.kfold != fold].reset_index(drop = True)
#####################
後略
#####################上記の変更をしないままに書籍のコードをそのまま実行すると、以下のエラーが出てきました。
Traceback (most recent call last):
File "/Users/XXXX/Desktop/programming/approaching_any_mlp/categorical/src/ohe_logres.py", line 63, in <module>
run(fold_)
File "/Users/XXXX/Desktop/programming/approaching_any_mlp/categorical/src/ohe_logres.py", line 53, in run
model.fit(x_train, df_train.income)
File "/Users/XXXX/miniconda3/envs/almost_ml/lib/python3.12/site-packages/sklearn/base.py", line 1151, in wrapper
return fit_method(estimator, *args, **kwargs)
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/XXXX/miniconda3/envs/almost_ml/lib/python3.12/site-packages/sklearn/linear_model/_logistic.py", line 1215, in fit
check_classification_targets(y)
File "/Users/XXXX/miniconda3/envs/almost_ml/lib/python3.12/site-packages/sklearn/utils/multiclass.py", line 215, in check_classification_targets
raise ValueError(
ValueError: Unknown label type: unknown. Maybe you are trying to fit a classifier, which expects discrete classes on a regression target with continuous values.
最終行で言っていることは、「ロジスティック回帰分析は目的変数が離散的なクラス(discrete classes)じゃなきゃいけないんだけど、連続値(continuous values)かよくわからん値入れてない?」と言われてしまっています。
実際に「print(df.income.dtype)」を実行して目的変数incomeのデータ型を確認すると、「object」と返ってきたので、離散的な値として認識されていなかったことが分かります。
なので、「df.income.astype(int)」で置き換えてあげることによって、離散値(=整数)であることを明示しています。
同様の変更はP124のXGBOOSTを実行する時も施します。
def run(fold):
#load ...
df = pd.read_csv('../input/adult_folds.csv')
#####################
中略
#####################
for col in features:
df.loc[:, col] = df[col].astype(str).fillna('NONE')
# 以下のコードを挿入
df.income = df.income.astype(int)
# now ...
for col in features:
#####################
後略
#####################それ以降も度々df.incomeが出てくる場面があるので、適宜int型に変換してください。
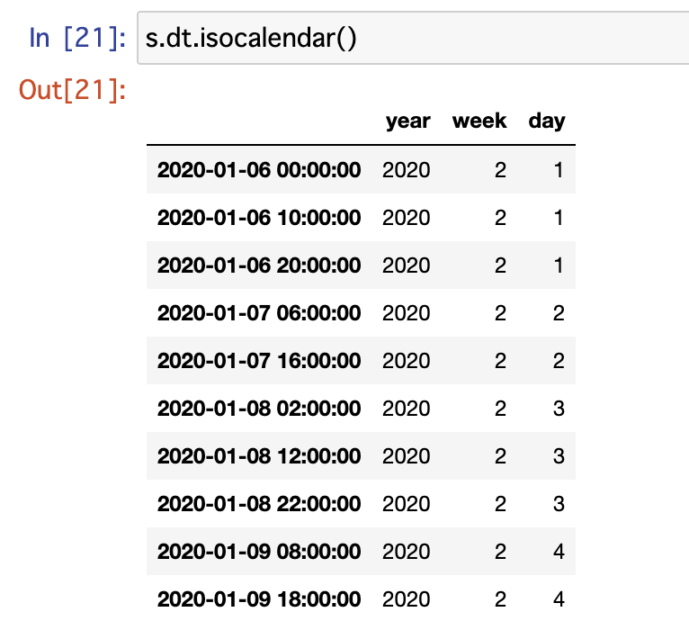
P142のweekofyearが使えない問題
修正版のコードは以下の通りになります。
import pandas as pd
s = pd.date_range('2020-01-06', '2020-01-10', freq = '10H').to_series()
features = {
'dayofweek': s.dt.dayofweek.values,
'dayofyear': s.dt.dayofyear.values,
'hour': s.dt.hour.values,
'is_leap_year': s.dt.is_leap_year.values,
'quarter': s.dt.quarter.values,
'weekofyear': s.dt.isocalendar().week.values
}現在のPandas(2024年1月現在の安定版は2.2.0)ではpandas.Series.dt.weekofyearは使えないので、代わりにpandas.Series.dt.isocalendar()でyear, week, dayをカラムに持つDataFrameからweek列を持ってくることで同様の機能を実現します。
P174のPipeline作成
修正版のコードは以下の通りです。
######################
前略
######################
if __name__ == '__main__':
# Load ...
train = pd.read_csv('../input/train.csv')
# 以下のコードを追加
test = pd.read_csv('../input/test.csv')
# we ...
idx = test.id.values.astype(int)
###################
後略
###################純粋なtestデータの読み込み忘れですね。
P178のベイズ最適化
修正版のコードは以下の通りです。
######
前略
######
def optimize(params, param_names, x, y):
params = dict(zip(param_names, params))
######
中略
######
optimization_function = partial(
optimize,
param_names = param_names,
x = X,
y = y
)
# 以下のコードを追加
np.int = int
result = gp_minimize(
optimization_function,
dimensions = param_space,
n_calls = 15,
n_random_starts = 10,
verbose = 10
)
#####
後略
#####エラーの原因は、numpyの仕様が変わって、np.intという整数型の指定ができなくなったことに由来します。
にもかかわらず、scikit-optimize(skopt)の中のコードではastype(np.int)で整数型への変換を指定してしまっているので、それを「np.int = int」という一文でastype(int)に置き換えているわけです。
以下が修正前のコードを実行した際のエラーコードです。
Traceback (most recent call last):
File "/Users/username/Desktop/programming/approaching_any_mlp/categorical/src/rf_gp_minimize.py", line 68, in <module>
result = gp_minimize(
^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/optimizer/gp.py", line 259, in gp_minimize
return base_minimize(
^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/optimizer/base.py", line 298, in base_minimize
next_x = optimizer.ask()
^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/optimizer/optimizer.py", line 367, in ask
return self._ask()
^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/optimizer/optimizer.py", line 434, in _ask
return self.space.rvs(random_state=self.rng)[0]
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/space.py", line 900, in rvs
columns.append(dim.rvs(n_samples=n_samples, random_state=rng))
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/space.py", line 158, in rvs
return self.inverse_transform(samples)
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/space.py", line 528, in inverse_transform
inv_transform = super(Integer, self).inverse_transform(Xt)
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/space.py", line 168, in inverse_transform
return self.transformer.inverse_transform(Xt)
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/transformers.py", line 309, in inverse_transform
X = transformer.inverse_transform(X)
^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/skopt/space/transformers.py", line 275, in inverse_transform
return np.round(X_orig).astype(np.int)
^^^^^^
File "/Users/username/miniconda3/envs/almost_ml/lib/python3.12/site-packages/numpy/__init__.py", line 324, in __getattr__
raise AttributeError(__former_attrs__[attr])
AttributeError: module 'numpy' has no attribute 'int'.
`np.int` was a deprecated alias for the builtin `int`. To avoid this error in existing code, use `int` by itself. Doing this will not modify any behavior and is safe. When replacing `np.int`, you may wish to use e.g. `np.int64` or `np.int32` to specify the precision. If you wish to review your current use, check the release note link for additional information.
The aliases was originally deprecated in NumPy 1.20; for more details and guidance see the original release note at:
https://numpy.org/devdocs/release/1.20.0-notes.html#deprecations. Did you mean: 'inf'?
P241のTfidfVectorizerのget_feature_name
修正版のコードは以下になります。
import pandas as pd
import nltk.tokenize import word_tokenize
######
中略
######
sample_index = 0
feature_scores = dict(
zip(
# 以下1コードを修正
tfv.get_feature_names_out()
corpus_svd.components_[sample_index]
)
)scikit-learnのTfidfVectorizerの仕様が変更され、特徴量の名前を取得するメソッドはget_feature_names()からget_feature_names_out()になった関係で以上のように修正しています。
P262~271のNLP
今回の修正では、config.pyとmodel.pyを修正します。
config.pyの修正は以下の通りです。
# config.py
import transformers
#### 中略 ####
TOKENIZER = transformers.BertTokenizer.from_pretrained(
BERT_PATH,
do_lower_case = True,
return_dict = False ## このコードを追加
)model.pyも同様に修正します。
# model.py
import config
import transformers
import torch.nn as nn
class BERTBaseeUncased(nn.Module):
def __init__(self):
super(BERTBaseUncased, self).__init__()
# we ...
# config.py
self.bert = transformers.BertModel.from_pretrained(
'bert-base-uncased', ## 参照する事前学習済みモデルを直接指定
return_dict = False ## config.pyと同様に追加
)
### 後略 ###なお、上記修正を加えないと以下の2つのエラーが出現します。
- HuggingFaceのモデルを持ってくるにしても、名前の指定の仕方がおかしいよ?エラー
- dropout()に入れる引数は文字列じゃなくてTensorを入れてくれる?エラー
# エラー1
huggingface_hub.utils._validators.HFValidationError: Repo id must be in the form 'repo_name' or 'namespace/repo_name': '../input/bert_base_uncased/'. Use `repo_type` argument if needed.
# エラー2
TypeError: dropout(): argument 'input' (position 1) must be Tensor, not str1つ目のエラーはconfig.pyのBERT_PATHが以下のように記載されていることによるものなので、多少強引ですが使いたい学習済みモデルを直接指定して解決しました。
本当はHuggingFaceから学習済みモデルbert-base-uncasedをダウンロードして当該フォルダに配置するのかもしれませんが、書籍にも記載がないので少し横着したやり方にはなっています。
# config.py
### 前略 ###
# define ...
BERT_PATH = '../input/bert_base_uncased/'2つ目のエラーはHugging Faceのtransformersライブラリがv3からv4に変更された際に起こるエラー(元々のコードはv3で書かれている)ので、return_dict = Falseを書き加えてライブラリの変更に対応しています。



コメント